Fixation de l’azote chez la gammaprotéobactérie diazotrophique marine largement répandue Candidatus Thalassolituus haligoni
Résumé
La diversité et la distribution mondiale des bactéries diazotrophes hétérotrophes (HBD) dans l’océan sont récemment devenues évidentes. Cependant, la compréhension des taux de fixation de diazote (N2) pour ces micro-organismes en grande partie non cultivés pose un défi majeur en raison de leurs exigences de croissance non définies et de la régulation complexe de l’enzyme nitrogénase. Nous avons isolé et caractérisé Candidatus Thalassolituus haligoni. L’analyse de son gène nifH par séquençage d’amplicon a révélé la vaste distribution de Cand. T. haligoni dans les océans Pacifique, Atlantique et Arctique. Les échantillons ont été collectés lors d’études de terrain menées le long des transects GEOVIDE (GEOTRACES GA01) à travers l’Atlantique Nord, canadien ArcticNet (GEOTRACES, GN03) à travers la baie de Baffin, sur le plateau néo-écossais et au niveau de l’archipel arctique canadien. Grâce à des approches combinées de génomique, de protéomique et de physiologie, nous avons confirmé que l’isolat fixe le N2, bien que les facteurs de la régulation de la nitrogénase chez Cand. T. haligoni restent peu clairs. Actuellement, seules deux cultures isolées avec des génomes entièrement séquencés sont connues dans le clade des Oceanospirillales, Parathalassolituus penaei (régionalement limitée) et Cand. T. haligoni (distribution mondiale). Compte tenu de son abondance, sa distribution, sa cultivabilité et la disponibilité de son génome, nous proposons Cand. T. haligoni comme modèle pour les γ-protéobactéries HBD, en particulier du clade des Oceanospirillales.
En outre, comme la fixation de N2 chez Cand. T. haligoni a lieu en présence de nitrate, nous proposons que le clade des diazotrophes Oceanospirillales puisse être un contributeur majeur au budget marin global de fixation de N2.
Figure
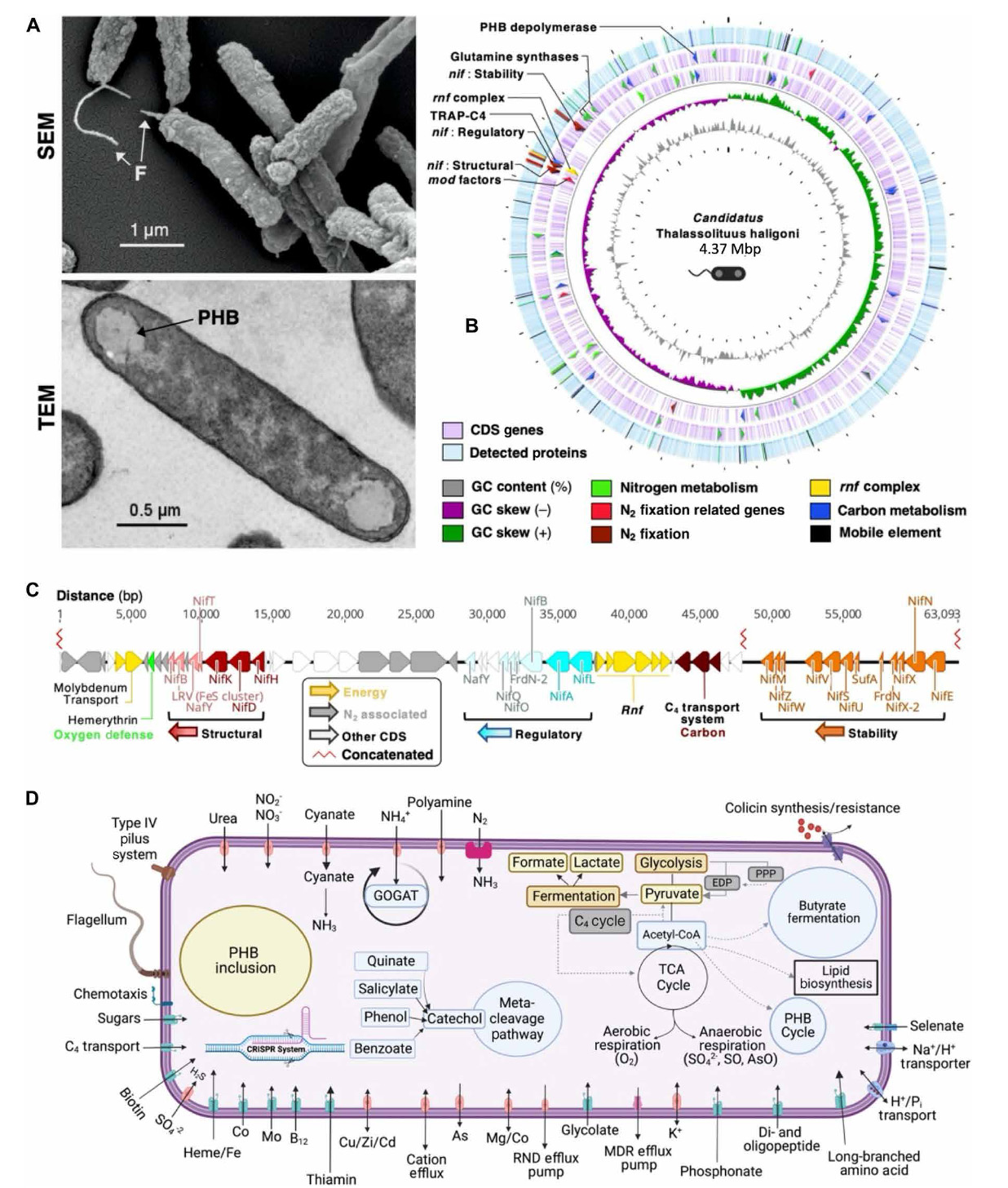
Fig. 1. Microscopie électronique à balayage et modèles métaboliques génomiques, protéomiques et assistés par le génome de Cand. T. haligoni.
(A) morphologie cellulaire de l’isolat dans des conditions de fixation de N2 par microscopie électronique à balayage (SeM ; en haut) et microscopie électronique à transmission (teM ; en bas). Grossissement SeM = 17,71 × 103. Les flèches indiquent les flagelles (F) et les granules PhB (PhB).
(B) Carte circulaire protéome-génome dans des conditions appauvries en NO3. En partant de l’anneau le plus interne : Contenu en GC (gris), biais de GC pour les brins avant (vert) et arrière (magenta), CDS des brins avant et arrière (violet clair), et protéines détectées (bleu). Les flèches dans le génome indiquent les gènes d’intérêt [clusters nif, les gènes liés à la fixation N2 sont l’absorption de molybdate et les gènes de transformation (facteurs mod), l’absorption de tRAP-c4, et la synthèse de glutamine] et les protéines détectées correspondantes sont mises en évidence dans les couleurs respectives. Les espaces blancs dans le protéome indiquent les régions non détectées.
(C) Clusters Nif (rouge, bleu et orange) et gènes de fixation N2 associés (gris). Les gènes associés à la fixation N2 comprennent les gènes de glycogène phosphorylase et d’absorption du glycogène. les nombres le long de la ligne indiquent les emplacements des paires de bases dans le génome. Une rupture nucléotidique au centre du diagramme entre 3 598 007 nt et 3 737 467 nt est représentée par la ligne en zigzag en rouge (concaténée). Les clusters sont codés par couleur en fonction des clusters nif associés, les gènes accessoires de ce cluster étant indiqués par des teintes de couleur respectives.
(D) Schéma métabolique simplifié de l’isolat basé sur l’annotation du génome. Le schéma a été créé à l’aide du serveur RASt et de BioRender.
Référence
e. « Nitrogen fixation in the widely distributed marine γ-proteobacterial diazotroph Candidatus Thalassolituus haligoni« . Science Advances Volume 10, Issue 31 (Aug 2024)